Gensaxar är ett samlingsnamn för tekniker som används för att redigera DNA. Det kallas för genomredigering (även genredigering eller geneditering). Det mest använda tekniken är CRISPR/Cas9.
CRISPR/Cas9 – i korthet
Populärt kallas CRISPR/Cas9 för gensaxen. Det beror på att den har ett enzym, Cas9, som kan klippa av den dubbelsträngade DNA-molekylen. Vart den klipper går att styra, det är just det som gör CRISPR/Cas9 till en så användbar och revolutionerande teknik.
Klippet utförs vid den plats i DNA-molekylen som ett guide-RNA binder till. Det kan vara en gen som forskaren är intresserad av att ändra eller stänga av. Guide-RNA:t designas innan experimentet och designas om när platsen för redigeringen ändras. Ett guide-RNA är vägvisare till Cas9.
Efter att Cas9 klippt av DNA-spiralen inne i cellkärnan lagar cellens reparationssystem dubbelsträngsbrottet. Att skadan lagas snabbt är viktigare än att den blir perfekt och ofta uppstår en genetisk förändring, en mutation, i lagningen.
Med CRISPR/Cas9 kan alltså nya mutationer induceras på ett förutbestämt ställe i DNA.
CRISPR/Cas9 är ursprungligen en försvarsmekanism hos bakterier som utvecklats till en genteknik. Det finns fler verktyg som kan användas till genomredigering, inte minst utvecklingar av CRIPSR/Cas9 och CRISPR-system med andra Cas-enzym. Här nedan kan du läsa om hur CRISPR/Cas9 fungerar i detalj, både som teknik och i bakterier.
Saxen och vägvisaren i CRISPR/Cas9
CRISPR/Cas9 består av två huvudkomponenter: ett guide-RNA och enzymet Cas9.
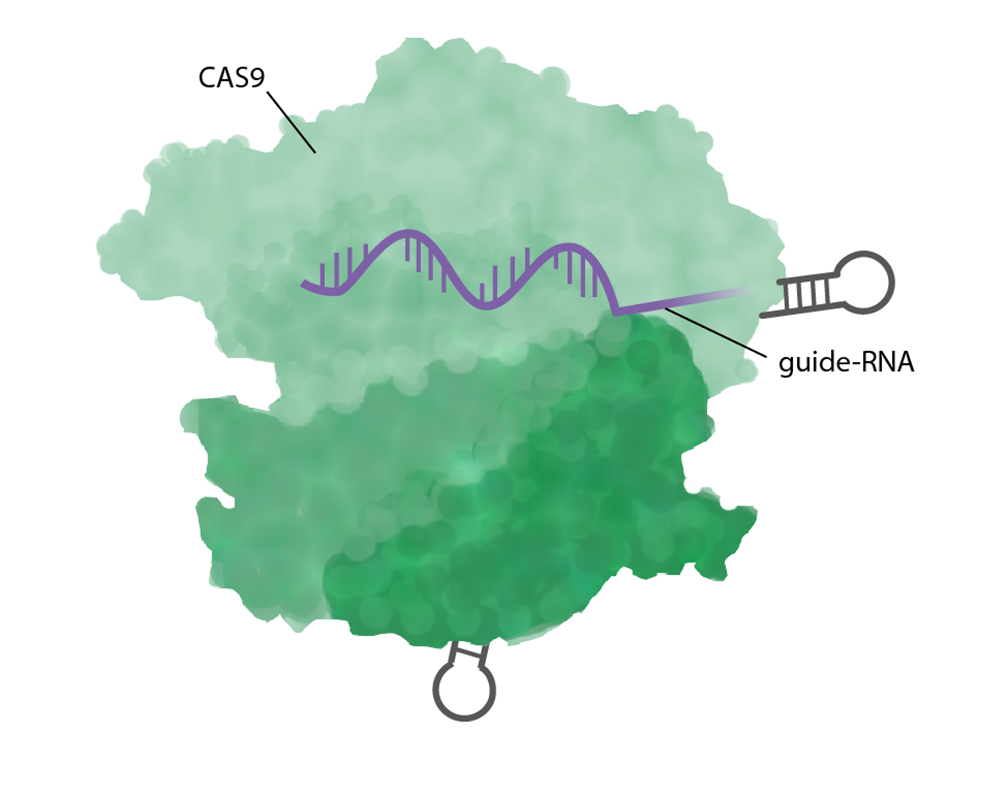
- Ett guide-RNA är en 20 baser kort RNA-sekvens som binder komplementärt (basparar) med den DNA-sekvens man vill ändra, till exempel en bit av en gen. Forskaren designar och låter tillverka ett nytt guide-RNA till varje unik redigering.
- Enzymet Cas9 är ett endonukleas som klipper i DNA. Det består av flera olika domäner som har olika funktioner.
Så här fungerar tekniken CRISPR/Cas9:
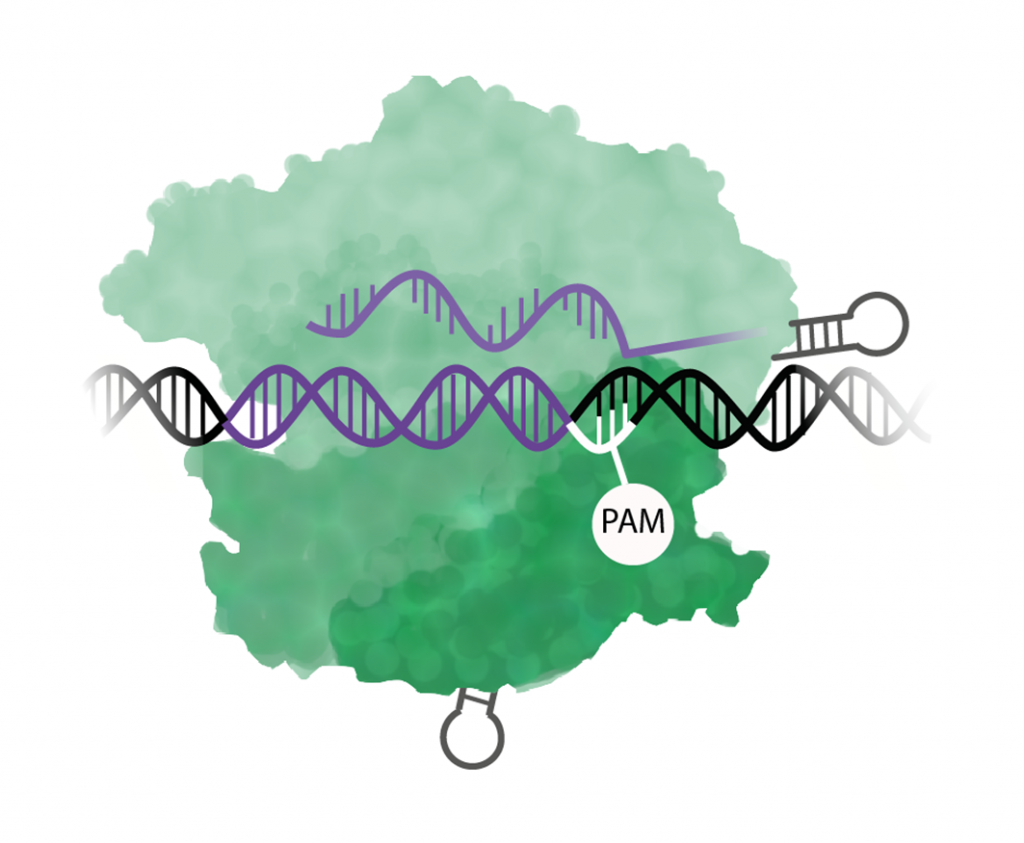
Steg 1. Cas9 känner igen PAM
Cas9 söker igenom DNA-molekylen efter ett särskilt motiv av tre baser som kallas för en PAM-sekvens (en förkortning av engelskans protospacer adjecent motif). För just Cas9 är PAM-sekvensen NGG vilket betyder att den första kan vara vilken som helst (A, C, T, G) och de två sista är GG. När en PAM kommer i DNA-sekvensen fungerar det som en stoppskylt och Cas9 stannar upp.
Steg 2: Cas9 öppnar upp DNA-spiralen
När Cas9 hittar en PAM-sekvens så används en av enzymets domäner till att öppna upp den dubbla DNA-strängen. Därefter försöker det guide-RNA som Cas9 är kopplat till att binda till (baspara) med DNA-sekvensen vid PAM.
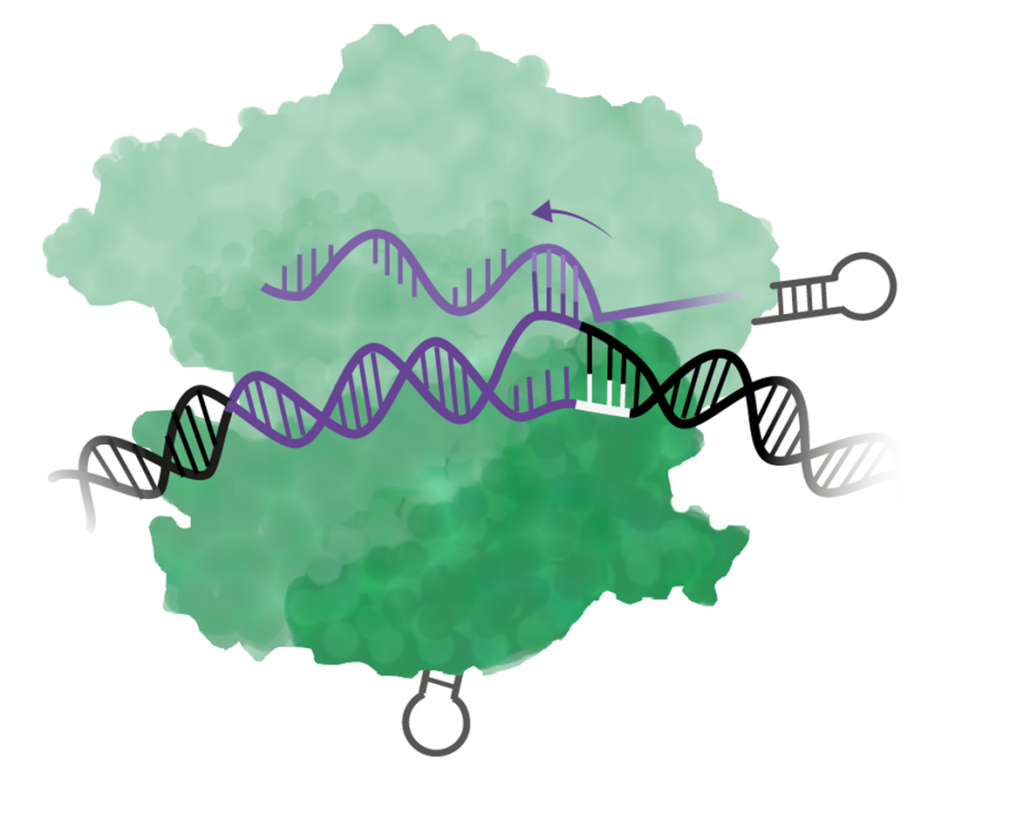
Steg 3: Alla 20 baser i RNA måste matcha DNA-sekvensen
Om inte alla baser i guide-RNA binder till DNA-sekvensen så släpper Cas9 och letar vidare efter nästa PAM.
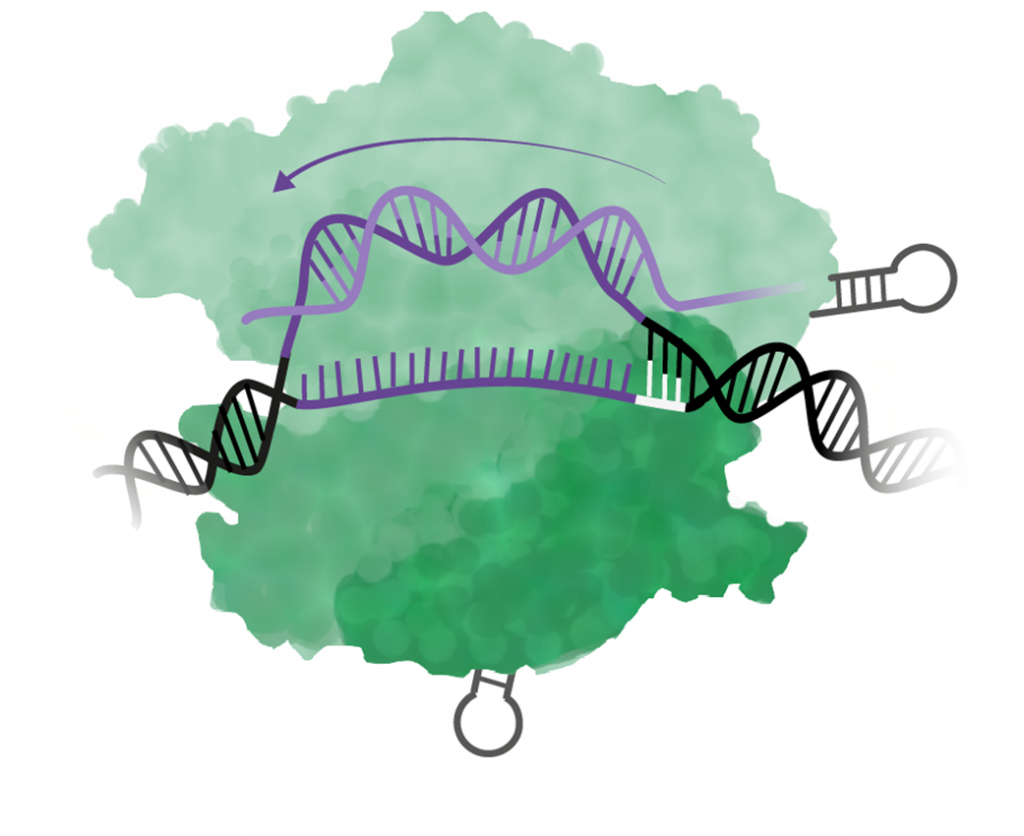
Steg 4: Cas9 klipper om allt stämmer
Om alla baser i guide-RNA binder till DNA-sekvensen så klipper Cas9 av båda DNA-strängarna, ett så kallat dubbelsträngsbrott. Klippet görs tre baser uppströms (5´) om PAM.
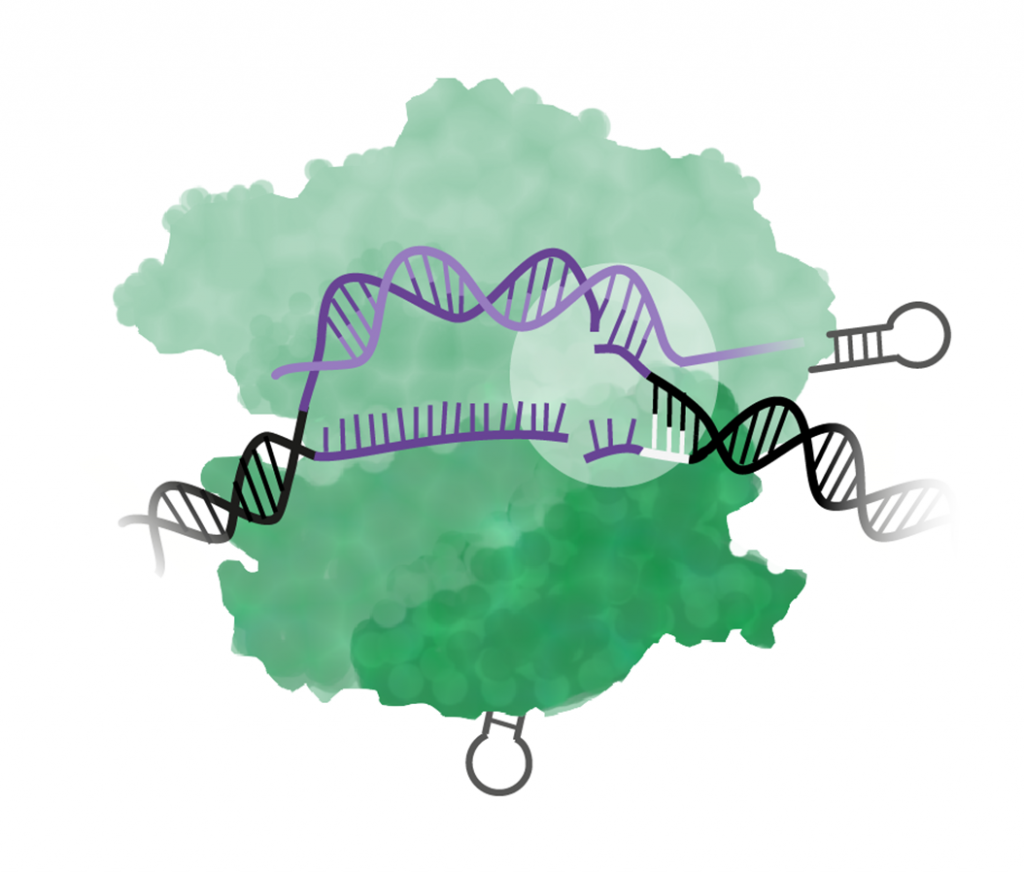
Steg 5. I lagningen av klippet uppstår en mutation
När DNA-strängarna skadas aktiveras cellens reparationssystem, men när lagningen inte blir perfekt uppstår en mutation – då sker själva genomredigeringen!
Celler har två olika reparationsmekanismer som i sin tur kan leda olika typer av mutationer: icke-homolog sammanfogning och homologistyrd reparation.
Den vanligaste reparationen vid ett dubbelsträngsbrott är icke-homolog sammanfogning (eng. non-homologous end-joining, NHEJ). Den här typen av reparation kan äga rum när som helst under cellcykeln och sammanfogar bara direkt de avbrutna DNA-strängarna. Om lagningen blir perfekt leder det till att Cas9 klipper av DNA-sekvensen än en gång, tills lagningen inte blir perfekt utan DNA-sekvensen förändras och en mutation har uppstått. Då matchar inte längre guide-RNA och Cas9 släpper taget.
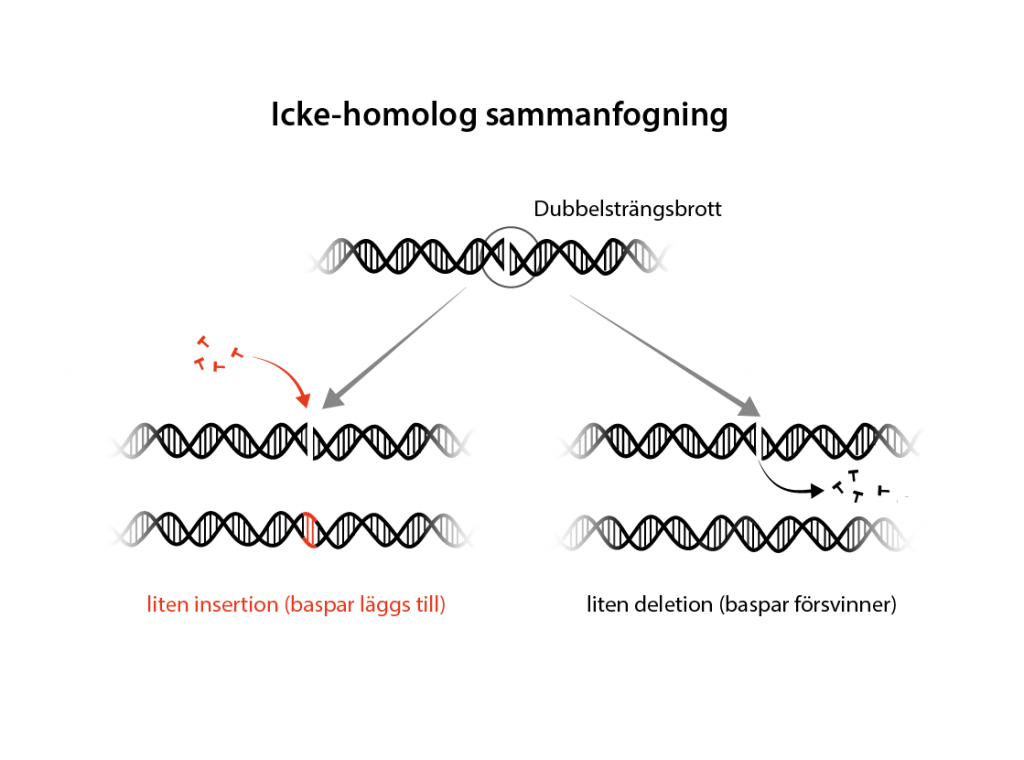
Mutationen som kan uppstår vid icke-homolog sammanfogning kallas för en insertion, om baspar tillkommer, eller en deletion om baspar försvinner. Om en insertion eller deletion uppstår i en gen blir konsekvensen att läsramen förskjuts åt något håll. Det i sin tur leder till att det i sekvensen snabbt uppkommer ett stoppkodon som avslutar transkriptionen och genen stängs av.
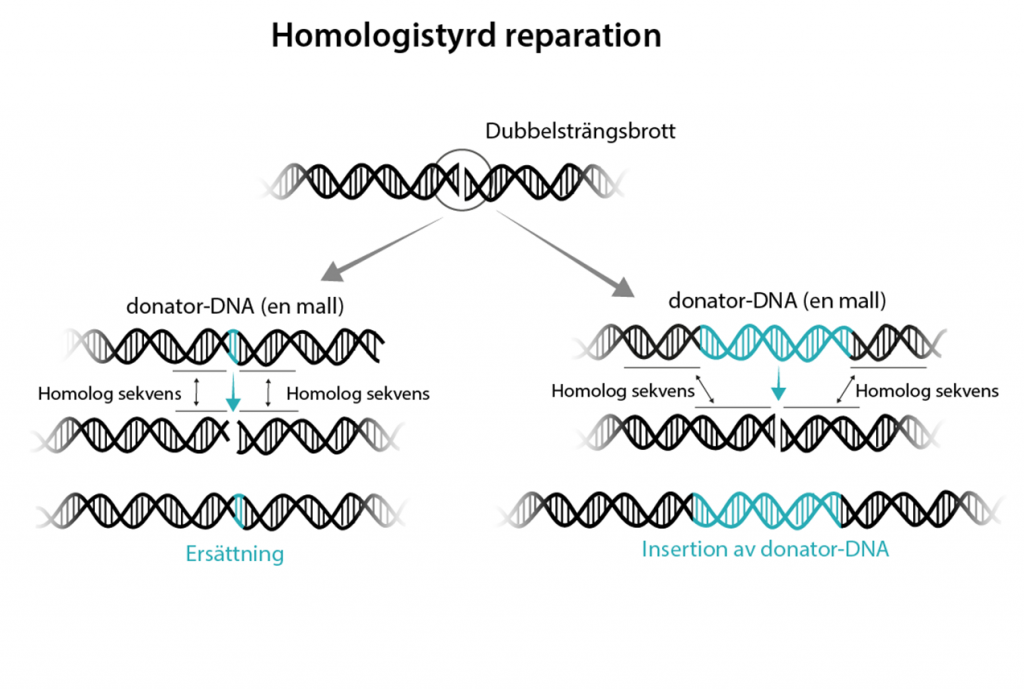
Den andra reparationsmekanismen kallas homologistyrd reparation (eng. homology directed repair, HDR) och är betydligt mindre frekvent. Här ges tillfälle för forskaren att föra in en ny DNA-sekvens så att resultatet av reparationen blir möjlig att styra. Homologistyrd reparation kan bara ske under visa faser av cellcykeln (G2 och S-fasen) så det gäller att optimera förhållandena i cellen samt tillsätta en mall till lagningen som kallas för donator-DNA.
Så här fungerar CRISPR/Cas i bakterier
Förlagan till CRISPR/Cas9 finns naturligt hos vissa bakterier och arkeér där det fungerar som ett försvar mot virus. De virus som infekterar bakterier och arkeér kallas för bakteriofager, eller bara fager. Bakteriofager fäster till bakteriens utsida och injicerar sitt DNA (eller RNA om RNA-virus) vilket leder till att proteintillverkningen i den infekterade cellen ställs om och börjar tillverka virusets proteiner. De nya proteinerna sätts samman till nya virus som kan spridas vidare.
En försvarsmekanism mot virus som upptäckts i bakterier och arkeér är CRISPR/Cas. I korthet så identifierar CRISPR/Cas främmande DNA från virus inne i cellen och klipper sönder det innan proteintillverkningen ställts om. Det finns olika CRISPR/Cas-system med olika egenskaper och uppgifter. Till exempel så har de olika Cas-enzym med olika PAM-sekvenser och de kan också klippa på olika sätt.
CRISPR/Cas9-systemet upptäcktes i bakterien Streptococcus pyogenes är det mest studerade och välkända systemet eftersom det översatts till en populär genteknik.
Så här fungerar CRISPR/Cas i bakterier steg för steg:
Steg 1: Cas identifierar och fångar upp virus-DNA, en ”spacer”
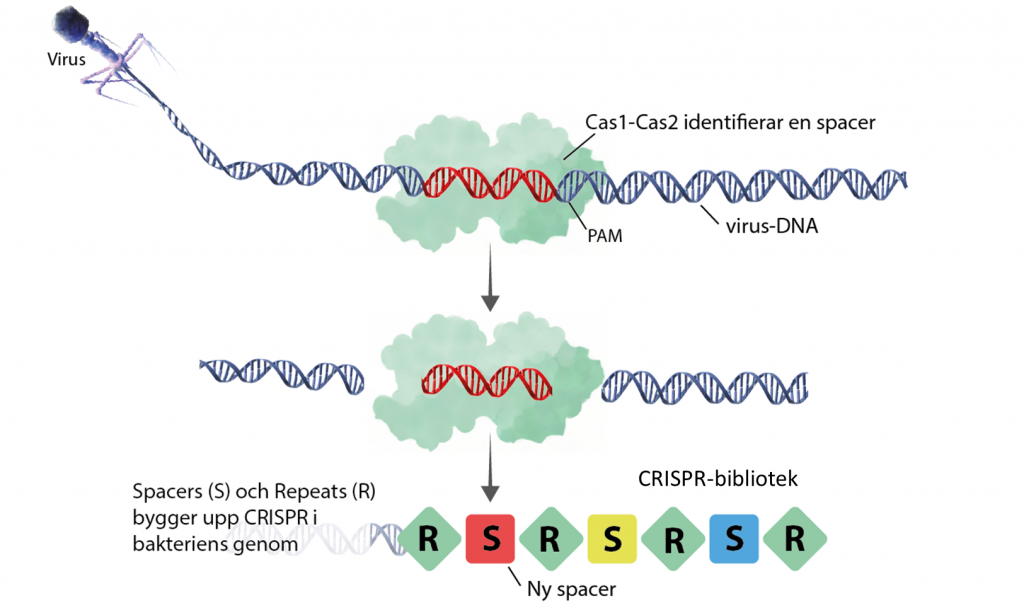
Första gången en bakterie infekteras av ett virus kan det upptäckas av två kopplade Cas-enzymer, Cas1 och Cas2. De letar efter DNA med sekvens-motiv som inte finns i bakteriens eget genom, så kallade PAM-sekvenser. När DNA från virus identifierats klipper Cas1 och Cas2 ut en liten bit DNA precis bredvid PAM-sekvensen och kilar in den i bakteriens eget genom som ett minne. Den lilla sekvensen virus-DNA kallas för en spacer.
Varje spacer (S) och omges av repeats (R). I en bakteries genom kan det finnas en till flera hundra spacers som bygger upp det genetiska minnes-bibliotek som kallas för CRISPR (Clustered regularly interspaced short palindromic repeats). När en bakterie delar sig och blir nya bakterier så följer CRISPR-biblioteket med.
Steg 2: crispr-RNA bildas
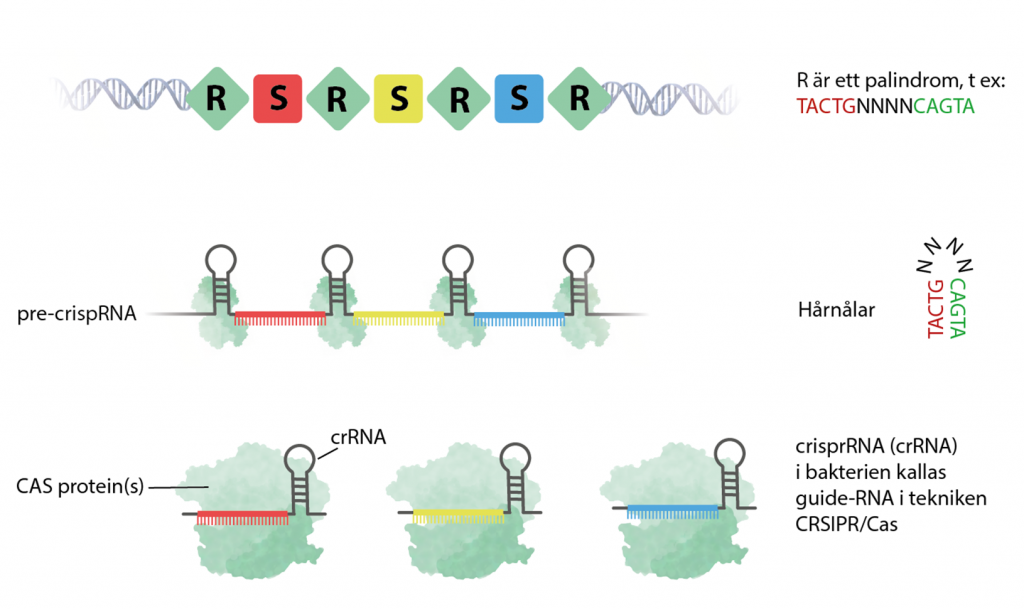
När CRISPR transkriberas bildas ett enda långt RNA som kallas pre–crisprRNA. Cas-enzymer klipper sedan upp det i mindre unika RNA-molekyler som kallas för crispr-RNA (cr(RNA), en molekyl för varje spacer.
De angränsande sekvenserna (R) är palindrom och översätts till den del av crispr-RNA som utgör ”hårnålen” eller ”öglan”. De består av några baser som efterföljs av baser som är komplementära till de första – fast baklänges. Hårnålens funktion är bland annat att ge crispr-RNA stabilitet.
Varje unikt crispr-RNA baseras på en spacer och känner igen ett visst sorts virus. När det kopplas samman med ett Cas, till exempel Cas9 så cirkulerar komplexet runt i bakterien i jakt på virus-DNA.
Steg 3: igenkänning och nedbrytning av virus-DNA
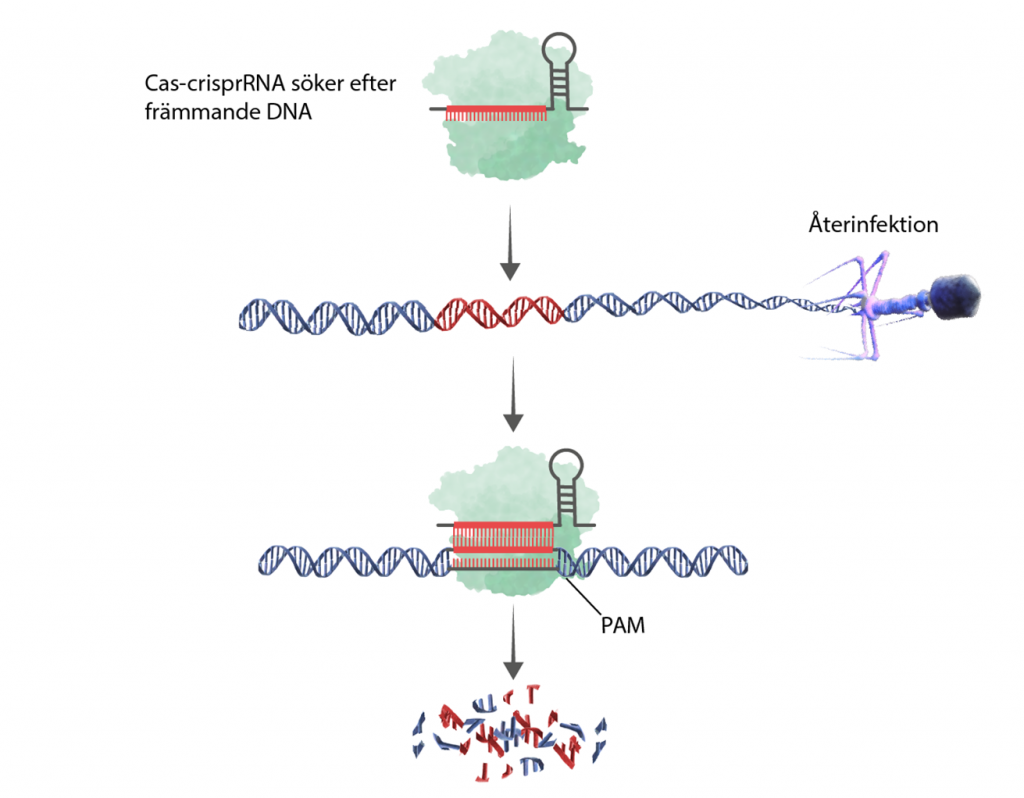
Cas letar efter en PAM-sekvens och öppnar upp DNA-spiralen för crispr-RNA som försöker baspara. Matchar crispr-RNA med DNA-sekvensen så är det virus-DNA, och då klipper Cas-enzymet. Olika Cas klipper olika, Cas9 gör till exempel ett klipp medan Cas3 fungerar mer som en gräsklippare som kör över och klipper sönder en längre sekvens. Resultatet är det samma: viruset elimineras.
Från bakterier till Nobelpris
Forskarna Jennifer Doudna och Emmanuelle Charpentier översatte den här försvarsmekanismen hos bakterier till en teknik för genomredigering. De ”upptäckte” inte CRISPR/Cas9 men såg potentialen och hittade de nycklar som krävdes för att få systemet att fungera även utanför bakterien. Doudna och Charpentiers samarbete såg ungefär ut så här:
Emmanuelle Charpentier studerade Streptococcus pyogenes, en bakterie som kan attackera blodceller och ge upphov till sjukdom och död. Hon upptäckte i bakterien en tidigare okänd molekyl, tracrRNA, som visade sig vara en del av CRISPR/Cas9. Ett tracrRNA kopplat till ett crisprRNA (crRNA) utgör tillsammans den igenkänningsmolekyl som leder Cas9 till virus-DNA. Charpentier publicerade upptäckten 2011. (I andra CRISPR/Cas system hos bakterier behövs inte tracr-RNA, därför finns det inte med i bilderna ovan).
Jennifer Doudna är en erfaren biokemist med stor kunskap om RNA. Samma år som Charpentier publicerade upptäckten om tracrRNA inledde de två ett samarbete. Tillsammans lyckades de få CRISPR/Cas9 att fungera även utanför bakterien, i ett provrör, och de förenklade de molekylära komponenterna så att den blev lättare att använda. De kopplade till exempel ihop tracrRNA och crRNA till en enda RNA-molekyl som de namngav guide-RNA.
I ett epokgörande experiment programmerade de om CRISPR/Cas9 som i sin naturliga form känner igen virus-DNA. Charpentier och Doudna visade att det går att styra den så att den klipper av vilken DNA-sekvens som helst- i nästan vilken organism som helst. När Cas9 klipper itu DNA försöker cellens reparationssystem laga skadan men lagningen blir sällan perfekt utan en eller ett par baser tillkommer eller försvinner. En mutation har uppstått!
Mutationen uppstår dessutom på ett förutbestämt ställe – tre baser uppströms om den PAM-sekvens som Cas9 känner igen. Charpentier och Doudna kunde alltså skriva om livets kod på ett mycket precist sätt. År 2020 fick de Nobelpriset i kemi för att ha utvecklat CRISPR/Cas9 till ett genomredigeringsverktyg som populärt brukar kallas för en gensax.
Andra CRISPR/Cas-system
Ungefär 50 % av alla bakterier, och 90 % av arkeér verkar ha någon form av CRISPR/Cas-system. CRISPR/Cas9 har utforskats och använts flitigast och har sitt ursprung i bakterien Streptococcus pyogenes – men det finns fler.
CRISPR/Cas12a kommer från bakterien Francisella novicida och skiljer sig från CRISPR/Cas9 på flera sätt. Det har till exempel en annan PAM-sekvens och ett mindre guide-RNA (endast crisprRNA och inte tracrRNA).
Ett annat exempel är CRISPR/Cas13 som finns naturligt i bakterien Leptotrichia shahii och som klipper i RNA istället för DNA. Nya endonukleas och CRISPR/Cas-system med andra egenskaper än CRISPR/Cas9 upptäcks kontinuerligt av forskare och verktygslådan utökas hela tiden med uppdaterade tekniker.
Andra tekniker för genomredigering
De första gensaxar som utvecklades kom på 1990-talet och baseras på enzymer som heter meganukleaser och zinkfingernukleaser (ZNF). Efter 2009 började även gensaxen TALEN användas och under det senaste decenniet har CRISPR/Cas9 totalt dominerat.
Liksom för CRISPR/Cas9 baseras TALEN och ZFN på nukleaser som klipper i DNA. Om ett nukleas klipper av DNA-spiralen i en levande cell så aktiveras cellens reparationssystem som lagar skadan. DNA-spiralen klistras ihop igen men ofta förändras DNA-sekvensen i snittet och en eller ett fåtal baser försvinner eller tillkommer. En mutation har då uppstått. Det går alltså att använda nukleaser för att inducera mutationer.
Riktigt användbara blir nukleaserna om det går att styra var i genomet de klipper och därmed var mutationen uppstår. Det kallas för att rikta mutationer och det är precis det som teknikerna används till. Ett nukleas måste då vägledas av en annan molekyl och dessa varierar för olika tekniker. För CRISPR/Cas9 är det till exempel ett guide-RNA som styr nukleset Cas9.
Meganukleaser
Meganukleaser är kompletta gensaxar som finns naturligt hos bakterier men även hos en del alger, jästsvampar och ett fåtal växter. De består av en del som känner igen och binder till en specifik DNA-sekvens, och en enzym-del som klipper itu samma sekvens. Flera hundra meganukleaser har upptäckts och renats fram, men de används inte i samma utsträckning som andra tekniker. Anledningen är att det har varit svårt för forskare att programmera om dem så att de binder till andra DNA-sekvenser än sina ursprungliga. Det innebär att antalet platser i genomet som kan redigeras är begränsade.
ZNF och TALEN
ZFN och TALEN är båda tekniker där ett nukleas satts ihop med en vägledande molekyl på konstgjord väg. Likt meganukleaser består de av två delar, en del som binder till DNA och en enzym-del som klipper itu DNA-spiralen. För varje ny genomredigering som en forskare vill göra så måste den DNA-bindande delen designas om för det specifika ändamålet. Sedan måste den delen sättas ihop med enzym-delen. ZNF och TALEN är mycket användbara gensaxar men något mindre flexibla än CRISPR/Cas9.
Vidareutvecklingar av CRISPR/Cas9
CRISPR/Cas9 är en ung metod och utvecklats hela tiden vidare främst eftersom nya enzymer upptäckts som har andra egenskaper än Cas9. Till exempel så kan Cas13 och Cas7-11 användas för att klippa i RNA istället för DNA. Det kommer väl till pass för forskare som studerar virus vars genom består av RNA och inte DNA. Ett sådant virus är sars-cov-2 som orsakar covid-19.
Ett annat enzym som heter Cas3 gör inte bara ett klipp i DNA-spiralen utan raderar en lång DNA-sekvens. Det kan vara användbart för forskare som studerar outforskade delar av genomet som inte innehåller gener. Vad händer när en region på en viss kromosom tas bort? Försvinner någon funktion?
Ytterligare en utveckling av gensaxen CRISPR/Cas9 är verktyget ”CRISPRoff” som kan stänga av och sätta på gener. I CRISPRoff är Cas9 utbytt mot ett protein som fäster en metylgrupp (CH3) intill en gen vilket gör att genen stängs av. Om metylgruppen sätts på igen så aktiveras genen åter. Du kan läsa mer om hur geners aktivitet, så kallat genuttryck, regleras med hjälp av bland annat metylgrupper på sidan Genuttryck.
I länkarna nedanför kan du läsa mer om hur genomredigering används:
- I USA har ett nötdjur med kortare päls avlats fram med hjälp av genomredigering och godkänts för marknaden. Syftet är att djuren ska tolerera hetta bättre. Mer information finns i forskningsnyheten: CRISPR-redigerade nötdjur har godkänts i USA.
- Ett svenskt företag har använt genomredigering för att förädla en potatissort med ändrad sammansättning av stärkelse. Syftet är att minska den kemiska bearbetning som används för att ta fram stärkelse. Mer information finns på sidan Amylopektinpotatis med CRISPR/Cas9.
- I USA har forskare tagit fram en medicinsk behandling som baseras på CRISPR/Cas9. Behandlingen är utvecklad för patienter med hemoglobinsjukdom och är den första i sitt slag som visats säker, ge behandlingseffekt och godkänts. Mer information i forskningsnyheten Världens första CRISPR-terapi godkänd inom EU.
- I Japan har en odlad matfisk med ökad muskelmassa tagits fram med genomredigering och godkänts för marknaden. Läs mer i forskningsnyheten Japan börjar sälja CRISPR-redigerad fisk.
- Japan har även godkänt två sorters tomater med förändrat näringsinnehåll som livsmedel. Mer information finns i forskningsnyheten CRISPR-redigerade tomater börjar säljas i Japan.
Uppdaterades 2024-06-25
